[SPSS] 등분산 가정이 성립하지 않는 일원 배치 분산 분석 (Welch's ANOVA, Brown-Forsythe ANOVA)
이전 포스팅에서 흡연 상태를 "1) 비흡연자", "2) 과거 흡연자", "3) 현재 흡연자"로 나누었고, 각 그룹의 폐기능 검사 중 하나인 FVC (Forced Vital Capacity)의 평균에 차이가 있는지 알아보고자 하여 일원 배치 분산 분석 (ANOVA)를 시행하였다. 2022.11.30 - [모평균 검정/SPSS] - [SPSS] 일원 배치 분산 분석 (One-way ANOVA, ANalysis Of VAriance)
ANOVA의 전제 조건은 두 가지였다.
1) 정규성
2) 등분산성
만약 '1) 정규성'은 만족하지만 '2) 등분산성'이 성립하지 않는 경우에는 어떻게 해야 할까?
당연히, 이전 포스팅의 일반적인 ANOVA는 실시할 수 없고, Welch's ANOVA 혹은 Brown-Forsythe ANOVA를 시행해야 한다.
- 이 Brown-Forsythe ANOVA는 Brown-Forsythe의 등분산성 검정과는 다른 것이다. (아래에서 설명함)
이번 포스팅에서는 이 Welch's ANOVA와 Brown-Forsythe ANOVA를 다뤄보고자 한다.
*실습용 데이터는 아래 링크를 클릭하면 다운로드할 수 있습니다.
2022.08.04 - [공지사항 및 소개] - 분석용 데이터 (update 22.11.28)
분석용 데이터 (update 22.11.28)
2022년 11월 28일 버전입니다. 변수는 계속하여 추가될 예정입니다. 다음 카테고리에 있는 글에서 이용된 데이터입니다. - 기술 통계 - 범주형 자료 분석 - 모평균 검정 - 반복 측정 자료 분석 - 통계
medistat.tistory.com
데이터를 불러오도록 한다. 불러오는 방법은 다음 링크를 확인하도록 한다.
2022.08.04 - [통계 프로그램 사용 방법/SPSS] - [SPSS] 데이터 불러오기 및 저장하기
목표: 거주 지역 단위에 따라 삶의 질의 평균이 모집단 수준에서 서로 다르다고 말할 수 있는가?
전제조건 (정규성)
Welch's ANOVA 및 Brown-Forsythe ANOVA에는 하나의 전제조건이 필요하다. 본 포스팅의 예시에 맞추어 설명하면 다음과 같다.
- 정규성: 대도시, 중소도시, 시골 거주자 별로 삶의 질의 분포를 보았을 때 각각의 분포는 정규성을 따른다.
그리고, 분산은 같지 않아도 된다.
2022.08.11 - [기술 통계/SPSS] - [SPSS] 정규성 검정
2022.08.18 - [기술 통계/SPSS] - [SPSS] 고급 Q-Q Plot - Van der Waerden, Rankit, Tukey, Blom
1) 정규성
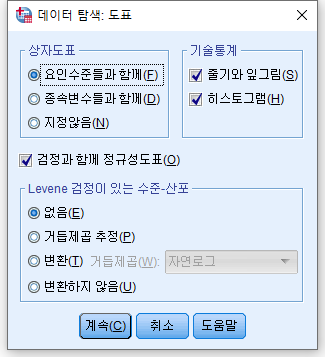
1) 분석(A) > 기술통계량(E) > 데이터 탐색 (E)
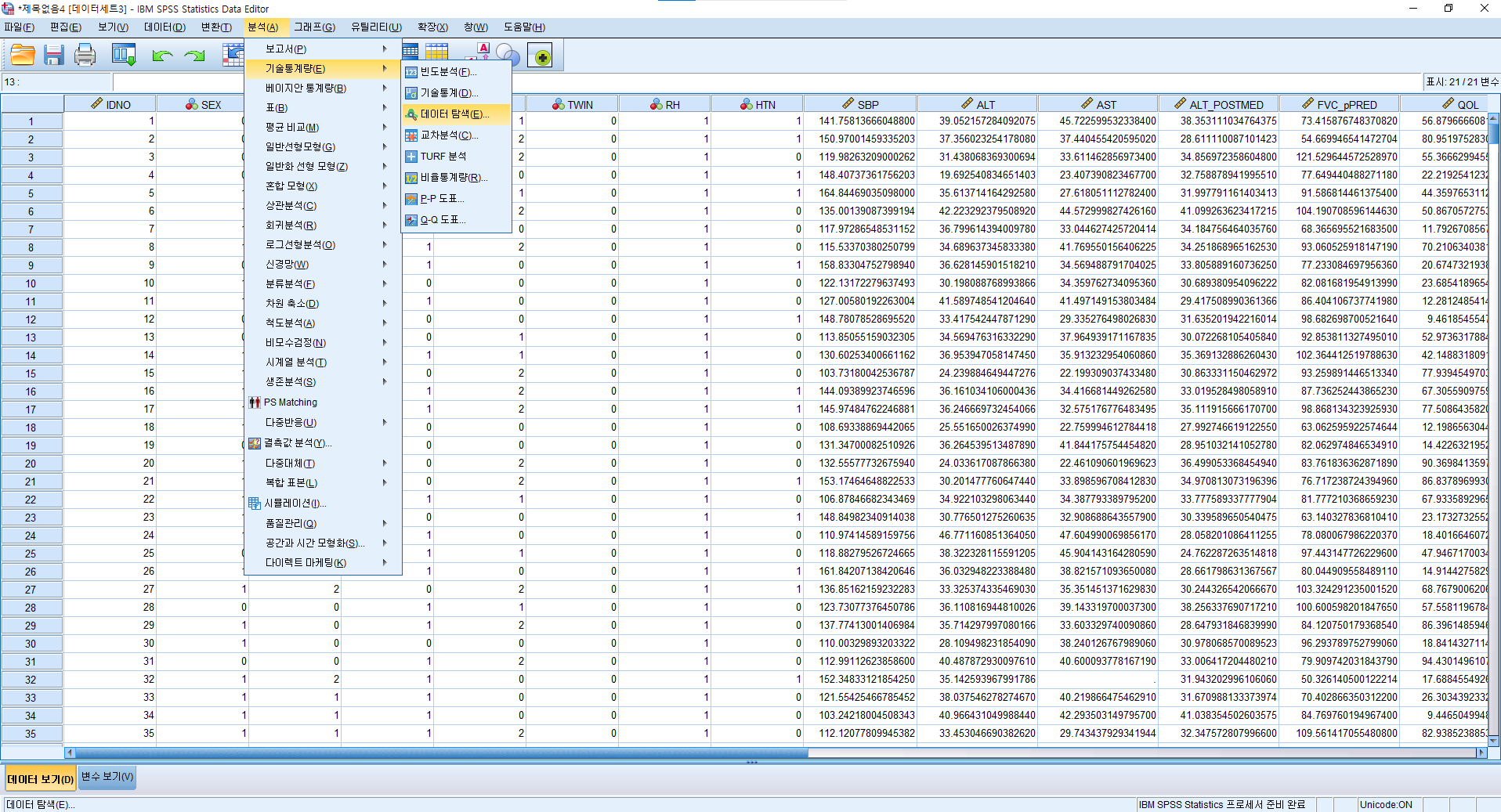
2) 분석하고자 하는 변수인 QOL을 "종속변수"에 넣어준다. 거주 지역 단위에 따라 정규성을 검정할 것이므로 "요인(F)"에 RESID를 넣는다. 그 뒤 "도표(T)..."를 선택한다.
3) "히스토그램(H)", "검정과 함께 정규성 도표(O)" 체크박스를 클릭하고 "계속(C)"를 누르고, 돌아가 "확인"을 누른다.
결과
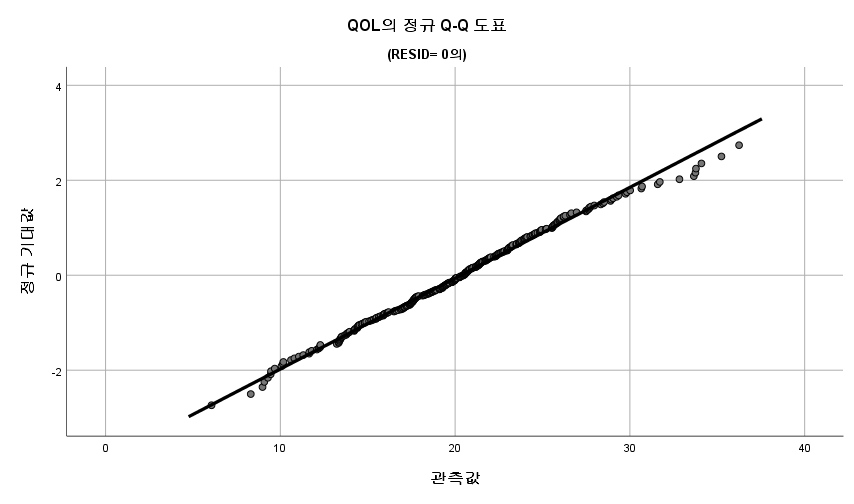
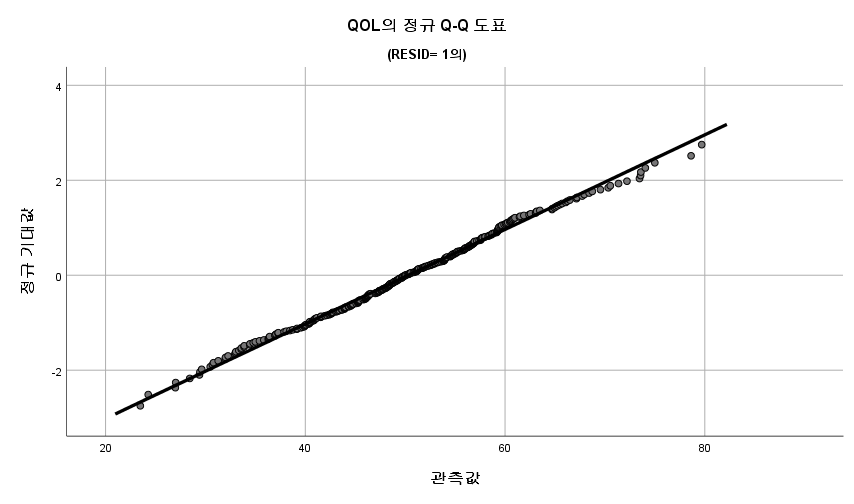
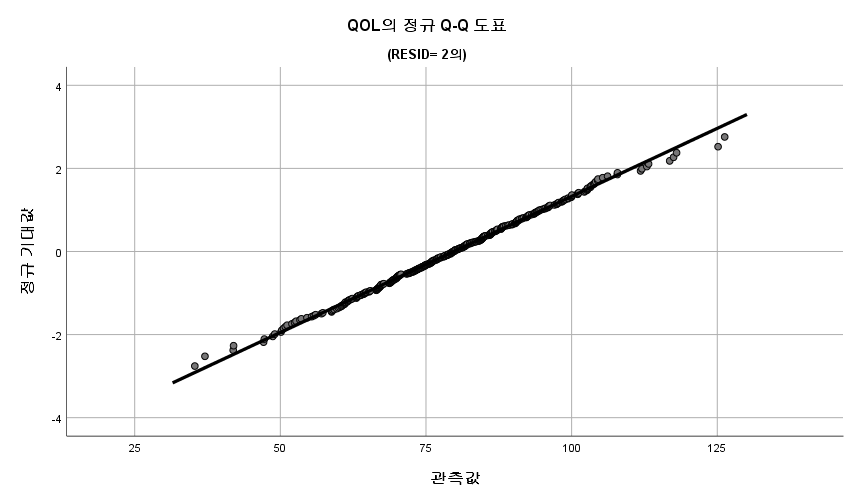
1) Q-Q plot
2) 히스토그램
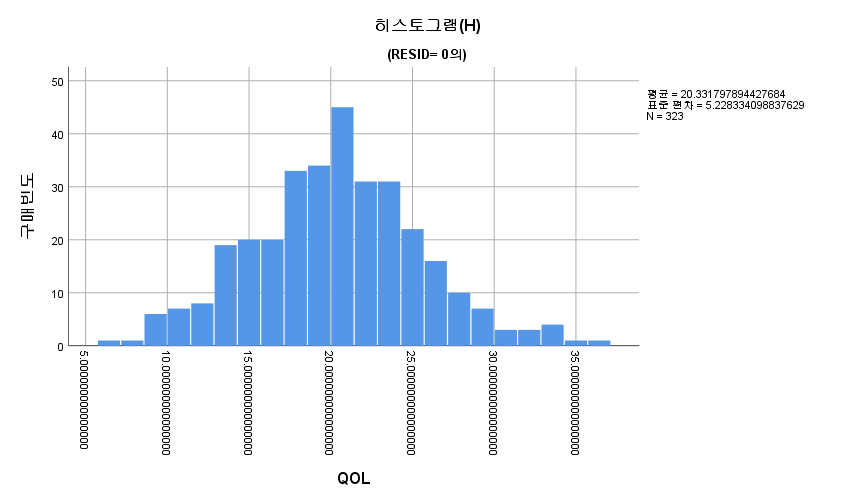
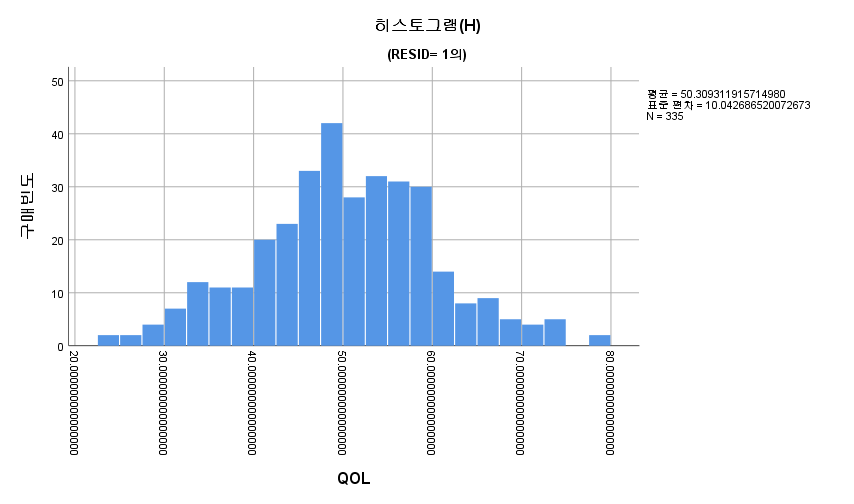
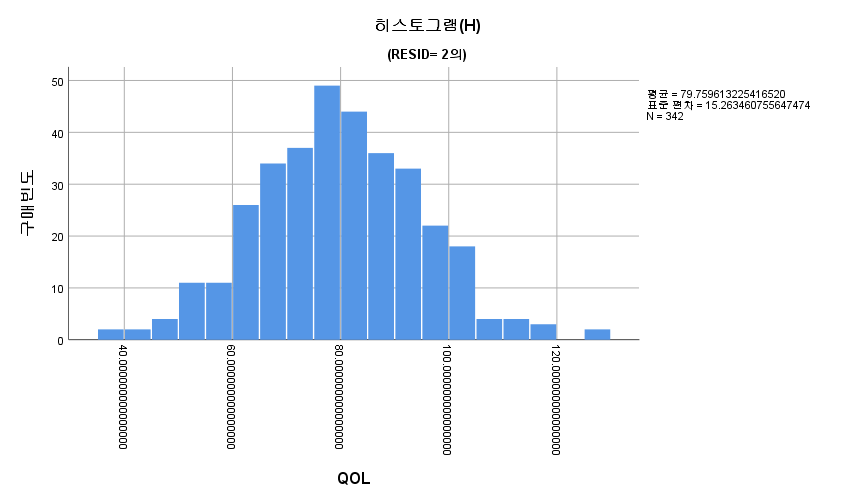
3) Shapiro-Wilk 검정
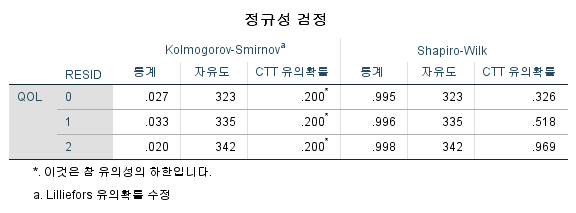
표본의 수가 2,000이 되지 않으므로 Shapiro-Wilk 통계량의 p-value를 보아야 하고, 이는 0.05보다 크다. Q-Q plot, 히스토그램에서 정규성을 띠지 않는다고 할만한 근거가 없으므로 정규성을 따른다고 결론을 짓는다.
따라서 정규성 전제는 따른다고 할 수 있다.
Welch's & Brown-Forsythe ANOVA 실행 방법
1) 분석(A) > 평균 비교 (M) > 일원배치 분산분석(O)
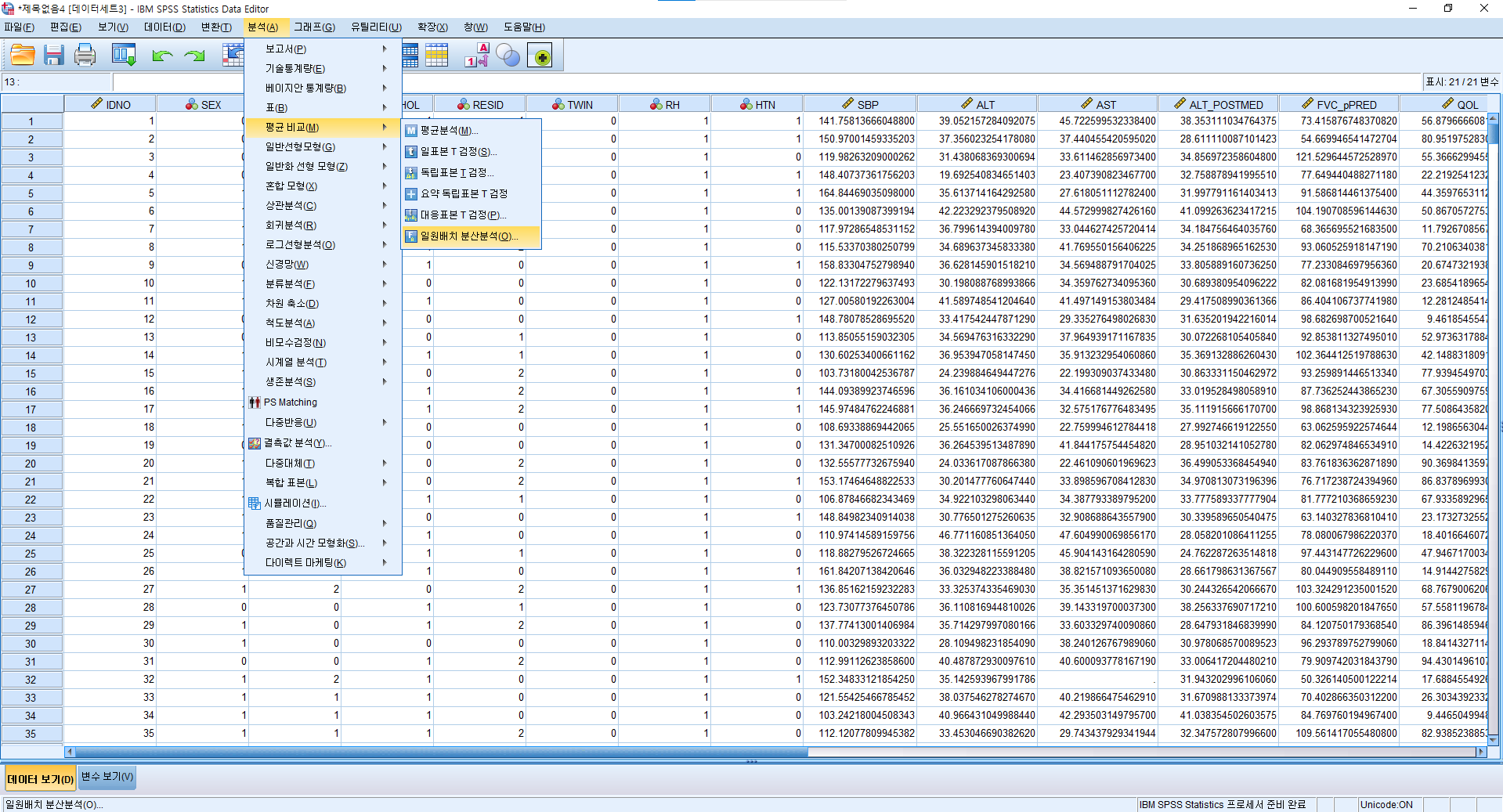
2) 분석하고자 하는 변수 QOL를 "종속변수(E)"쪽으로 옮기고, RESID을 "요인(F)"쪽으로 옮긴다. 그리고 "옵션"을 누른다.
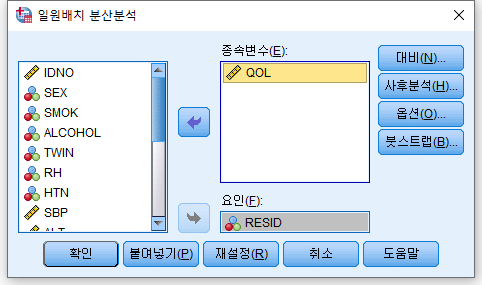
3) "분산 동질성 검정(H)" 체크박스를 선택한다. 분산이 같지 않을 경우를 대비해 "Brown-Forsythe", "Welch" 체크박스를 클릭한다. 그리고 "계속 (C)"을 누른다.
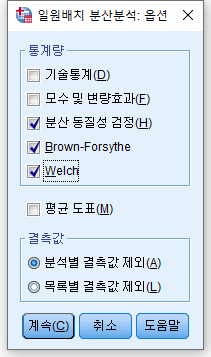
4) "사후분석(H)"을 클릭한다.
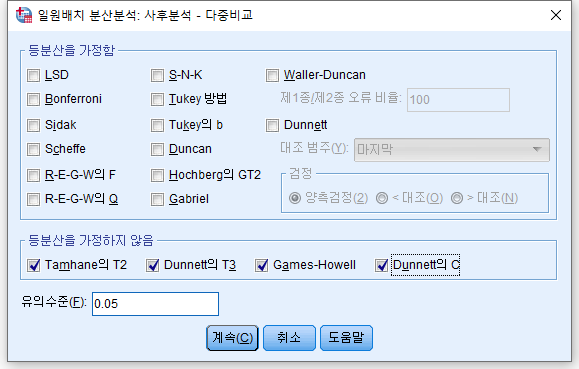
분산이 같지 않을 경우에만 대비하여 "Tanhame의 T2", "Dunnett의 T3", "Games-Howell", "Dunnett의 C"를 클릭한다. 분산이 같지 않은 경우 위쪽에 있는 사후 분석 방법은 사용할 수 없고 아래 사후 분석만 사용할 수 있다.
결과
1) 등분산성 검정
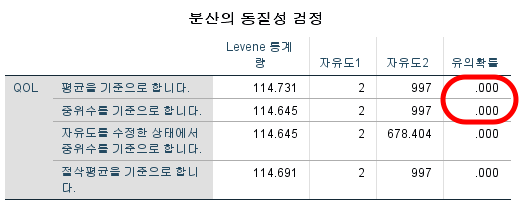
맨 위에 평균을 기준으로 한 검정이 일반적인 Levene의 등분산 검정이다.
그다음 중위수를 기준으로 한 검정이 Brown-Forsythe의 등분산성 검정이다.
어떤 것을 선택하는지는 개인의 선택이다. 하지만 이 경우 둘 다 0.05보다 작으므로 귀무가설을 기각하고, 분산에 차이가 있다고 결론 내린다.
2) 일반 ANOVA
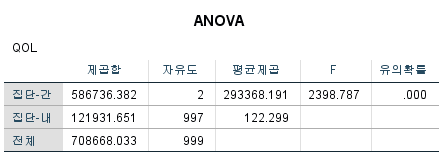
분산이 다르다고 결론 내렸으므로 이 표는 보면 안 된다.
3) Welch, Brown-Forsythe ANOVA
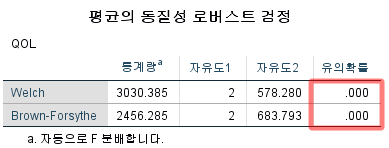
두 가지 방법 모두 p-value가 0.001보다 작다고 이야기하고 있다. 따라서 귀무가설을 기각하고 대립 가설을 채택한다. 그렇다면 여기에서 귀무가설 및 대립 가설은 무엇이었는가?
귀무가설: $H_0 =$세 집단의 모평균은 "모두" 동일하다.
대립 가설: $H_1 =$세 집단의 모평균이 모두 동일한 것은 아니다.
우리는 대립 가설을 채택해야 하므로 "세 집단의 모평균이 모두 동일한 것은 아니다."라고 결론 내릴 것이다.
그런데, 세 집단의 모평균이 모두 동일하지 않다는 말은 세 집단 중 두 개씩 골라 비교했을 때, 적어도 한 쌍에서는 차이가 난다는 것이다. 따라서 세 집단 중 두 개씩 골라 비교를 해보아야 하며, 이를 사후 분석 (post hoc analysis)이라고 한다. 세 집단에서 두 개씩 고르므로 가능한 경우의 수는$_3C_2=3$이다.
(1) 대도시 거주자 vs 중소도시 거주자
(2) 대도시 거주자 vs 시골 거주자
(3) 중소도시 거주자 vs 시골 거주자
사후 분석 결과를 볼 것이다.
이전 일반 ANOVA 포스팅(2022.11.30 - [모평균 검정/SPSS] - [SPSS] 일원 배치 분산 분석 (One-way ANOVA, ANalysis Of VAriance))에서는 여러 사후 분석 방법이 있다고 소개했었다. 하지만 등분산이 가정되지 않는 상황에서 쓸 수 있는 사후 분석 방법은 현저히 적어진다. 많이들 쓰는 방법은 다음 네 가지다.
1) Tamhane's T2
2) Games-Howell
3) Dunnett's T3
4) Dunnett's C
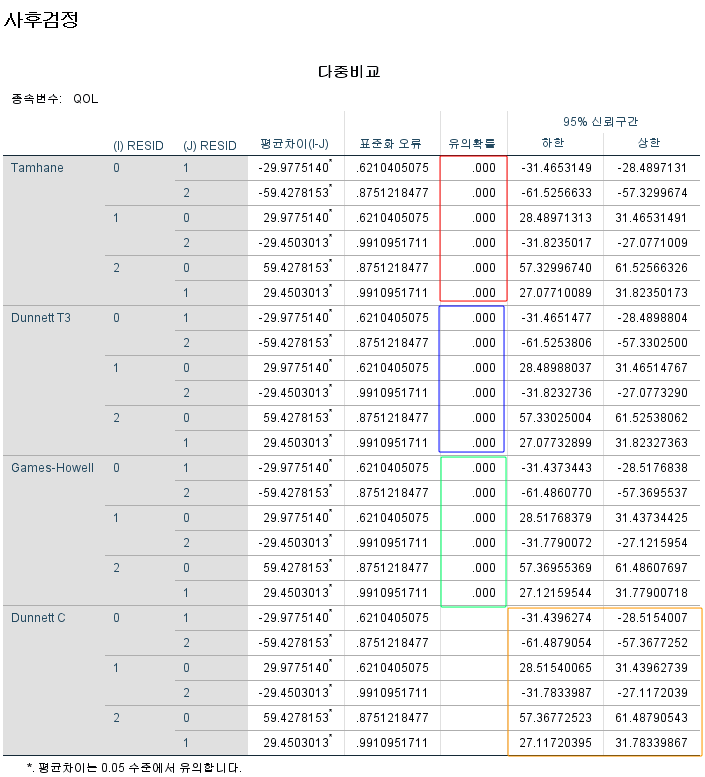
1) Tamhane's T2
2) Games-Howell
3) Dunnett's T3
4) Dunnett's C
박스 색깔과 검정방법을 매칭 시켜두었다.
위 표를 보는 법은 다음과 같다.
| 0 | 1 | 대도시 거주자 (0)와 중소도시 거주자 (1) 사이의 비교 |
| 2 | 대도시 거주자 (0)와 시골 거주자 (2) 사이의 비교 | |
| 1 | 0 | 대도시 거주자 (0)와 중소도시 거주자 (1) 사이의 비교 |
| 2 | 중소도시 거주자 (1)와 시골 거주자 (2) 사이의 비교 | |
| 2 | 0 | 대도시 거주자 (0)와 시골 거주자 (2) 사이의 비교 |
| 1 | 중소도시 거주자 (1)와 시골 거주자 (2) 사이의 비교 |
Dunnett C를 제외하고, 어떤 방법을 사용하든, 모든 비교에서 p-value는 0.001보다 작아 매우 유의하다. 그리고 Dunnett C는 신뢰구간에 0이 포함되지 않으면 유의한 것인데, 0을 포함하는 구간이 없다. 따라서 모든 비교에서 유의미한 FVC의 차이가 있다고 할 수 있다.
어떤 사후 분석을 쓸 것인가
이 논의에 대해 정답이 따로 있는 것은 아니다. 적절한 방법을 사용하여 논문에 제시하면 되고, 어떤 것이 정답이라고 콕 집어 이야기할 수는 없다. 다만, 사후 분석 방법이 여러 가지가 있다는 것은 '사후 분석 방법에 따라 산출되는 결과가 달라질 수 있다.'는 것을 의미하고, 심지어는 '어떤 사후 분석 방법을 채택하냐에 따라 유의성 여부가 달라질 수도 있다.'는 것을 의미한다. 심지어, Welch's or Brown-Forsythe's ANOVA에서는 유의한 결과가 나왔는데, 사후 분석을 해보니 유의한 차이를 보이는 경우가 없을 수도 있다. 따라서 어떤 사후 분석 방법 결과에 따른 결과인지 유의하여 해석할 필요가 있다.
[SPSS] 등분산 가정이 성립하지 않는 일원 배치 분산 분석 (Welch's ANOVA, Brown-Forsythe ANOVA) 정복 완료
작성일: 2022.11.30.
최종 수정일: 2022.11.30.
이용 프로그램: IBM SPSS v26
운영체제: Windows 10
'모평균 검정 > SPSS' 카테고리의 다른 글
| [SPSS] 윌콕슨 순위 합 검정, 맨 휘트니 U 검정 (비모수 독립 표본 중앙값 검정: Wilcoxon rank sum test, Mann-Whitney U test) (0) | 2022.12.03 |
|---|---|
| [SPSS] 일표본 윌콕슨 부호 순위 검정 (비모수 일표본 중앙값 검정: One-Sample Wilcoxon Signed Rank Test) (1) | 2022.12.02 |
| [SPSS] 일원 배치 분산 분석 (One-way ANOVA, ANalysis Of VAriance) (1) | 2022.11.30 |
| [SPSS] 독립 표본 T검정 (Independent samples T-test) (0) | 2022.11.30 |
| [SPSS] 일표본 T검정 (One-sample T-test) (1) | 2022.11.29 |