
[SAS] 등분산성 검정 (Homogeneity of variance) - PROC GLM
T-test, ANOVA 등 몇몇 분석에서는 분포들의 분산이 같다는 가정 (등분산성)이 필요하다. 이럴 때에는 등분산성 검정을 해야 하는데, 많이들 사용하는 방법은 크게 다섯 가지가 존재한다.
1) Fooled F
2) Levene
3) O'Brien
4) Brown and Forsythe
5) Bartlett
이 중 첫 번째 방법인 Fooled F 검정은 PROC TTEST에서 확인할 수 있다. 그리고 Levene의 등분산성 검정은 T-test에 관한 글에서 다루었으므로 본 포스팅에서는 다루지 않겠다. Fooled F와 Levene 등분산성 검정에 관한 내용은 다음 링크에서 확인할 수 있다. 2022.10.04 - [모평균 검정/SAS] - [SAS] 독립 표본 T검정 (Independent samples T-test) - PROC TTEST
결국 우리가 확인하고자 하는 것은, "수축기 혈압 분포의 분산이 고혈압 환자군과 일반인 사이에서 다르다고 할 수 있는가?"이다. 이에 대해 설면하도록 하겠다.
*실습용 데이터는 아래 링크를 클릭하면 다운로드할 수 있습니다.
2022.08.04 - [공지사항 및 소개] - 분석용 데이터 (update 22.08.29)
분석용 데이터 (update 22.08.29)
2022년 08월 29일 버전입니다. 변수는 계속하여 추가될 예정입니다. 다음 카테고리에 있는 글에서 이용된 데이터입니다. - 기술 통계 - 통계 프로그램 사용 방법 1) 엑셀 파일 2) CSV 파일 3) 코드북
medistat.tistory.com
시작하기 위해 라이브러리를 만들고, 파일을 불러온다.
라이브러리 만드는 방법: 2022.08.05 - [통계 프로그램 사용 방법/SAS] - [SAS] 라이브러리 만들기 - LIBNAME
파일 불러오는 방법: 2022.08.05 - [통계 프로그램 사용 방법/SAS] - [SAS] 데이터 불러오기 및 저장하기 - PROC IMPORT, PROC EXPORT
*라이브러리 지정하기;
LIBNAME hong "C:/Users/User/Documents/Tistory_blog";
*파일 불러오기;
PROC IMPORT
DATAFILE="C:\Users\user\Documents\Tistory_blog\Data.xlsx"
DBMS=EXCEL
OUT=hong.df
REPLACE;
RUN;
코드 - 등분산성 검정
등분산성을 검정하는 코드는 다음과 같다.
*O'Brien;
PROC GLM DATA=hong.df;
CLASS HTN;
MODEL SBP = HTN;
MEANS HTN / HOVTEST=OBRIEN(W=0.5);
RUN;
QUIT;
*Brown and Forsythe;
PROC GLM DATA=hong.df;
CLASS HTN;
MODEL SBP = HTN;
MEANS HTN / HOVTEST=BF;
RUN;
QUIT;
*Bartlett;
PROC GLM DATA=hong.df;
CLASS HTN;
MODEL SBP = HTN;
MEANS HTN / HOVTEST=BARTLETT;
RUN;
QUIT;PROC GLM DATA=hong.df; : 등분산 검정은 PROC GLM이라는 명령문으로 할 수 있다. PROC GLM을 시행하고, 데이터는 hong 라이브러리에 있는 df를 이용한다.
CLASS HTN; : 고혈압 여부(HTN)에 따라 분산이 같은지 검정할 것이다.
MODEL SBP = HTN; : 고혈압 여부(HTN)에 따라 수축기 혈압(SBP)의 분산이 같은지 검정할 것이다.
MEANS HTN / HOVTEST=OBRIEN(W=0.5); : 고혈압의 평균을 구해주어라. 등분산성 검정 (HOmogeneity of Variance TEST)은 O'Brien방법을 사용한다. O'Brien 방법의 parameter는 W가 있는데 원하는 값을 넣어주면 된다. 기본 값은 0.5로 설정되어 있으며, 기본값 그대로 사용하고자 할 때에는 "(W=0.5)"를 통째로 쓰지 않아도 괜찮다.
RUN; : 코드를 실행한다.
QUIT; : GLM코드는 QUIT구문을 실행하거나, 다른 코드를 실행해야만 끝나므로 QUIT을 실행한다.
Brown and Forsythe, Bartlett 방법은 각각 "OBRIEN"을 "BF", "BARTLETT"으로 바꾸어 구한 것이다.
결과
1) O'Brien 검정 결과
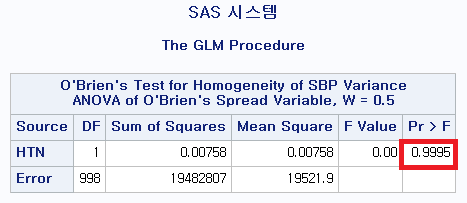
2) Brown and Forsythe 검정 결과
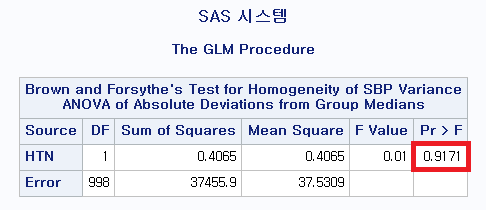
3) Bartlett 검정 결과

약간의 차이들은 있지만 모두 p-value가 0.05 이상으로 귀무가설을 기각하지 못해 모분산이 서로 같다고 결론 내릴 수 있다.
코드 정리
*라이브러리 지정하기;
LIBNAME hong "C:/Users/User/Documents/Tistory_blog";
*데이터 불러오기;
PROC IMPORT
DATAFILE="C:\Users\user\Documents\Tistory_blog\Data.xlsx"
DBMS=EXCEL
OUT=hong.df
REPLACE;
RUN;
*O'Brien;
PROC GLM DATA=hong.df;
CLASS HTN;
MODEL SBP = HTN;
MEANS HTN / HOVTEST=OBRIEN(W=0.5);
RUN;
QUIT;
*Brown and Forsythe;
PROC GLM DATA=hong.df;
CLASS HTN;
MODEL SBP = HTN;
MEANS HTN / HOVTEST=BF;
RUN;
QUIT;
*Bartlett;
PROC GLM DATA=hong.df;
CLASS HTN;
MODEL SBP = HTN;
MEANS HTN / HOVTEST=BARTLETT;
RUN;
QUIT;
SAS 등분산성 검정 (Homogeneity of variance) 정복 완료!
작성일: 2022.10.05.
최종 수정일: 2022.10.05.
이용 프로그램: SAS v9.4
운영체제: Windows 10