[SPSS] 윌콕슨 부호 순위 검정 (비모수 짝지어진 표본 중앙값 검정: Wilcoxon signed rank test)
짝지어진 두 분포의 평균이 다른지 확인하는 방법을 이전에는 대응 표본 T검정 (Paireed Sample T test)로 시행했었다. (2022.11.30 - [반복 측정 자료 분석/SPSS] - [SPSS] 대응 표본 T검정 (Paired samples T-test)) 하지만 여기에는 중요한 가정이 필요한데, 두 변수의 차이가 정규분포를 이룬다는 것이다. 하지만 차이의 분포가 정규성을 따르지 않는다면 어떻게 해야 할까? 그럴 때 사용하는 것이 Wilcoxon signed rank test (윌콕슨 부호 순위 검정)이다.
*실습용 데이터는 아래 링크를 클릭하면 다운로드할 수 있습니다.
2022.08.04 - [공지사항 및 소개] - 분석용 데이터 (update 22.12.01)
분석용 데이터 (update 22.12.01)
2022년 12월 01일 버전입니다. 변수는 계속하여 추가될 예정입니다. 다음 카테고리에 있는 글에서 이용된 데이터입니다. - 기술 통계 - 범주형 자료 분석 - 모평균 검정 - 반복 측정 자료 분석 - 통계
medistat.tistory.com
데이터를 불러오도록 한다. 불러오는 방법은 다음 링크를 확인하도록 한다.
2022.08.04 - [통계 프로그램 사용 방법/SPSS] - [SPSS] 데이터 불러오기 및 저장하기
목표: 모집단에서 GGT와 간기능 개선제 복용 후 GGT 중앙값에 차이가 있다고 할 수 있는가?
이번 포스팅의 목적은 1000명의 데이터를 가지고, 이 1000명이 기원한 모집단에서 GGT와 간 기능 개선제 복용 후 GGT의 중앙값에 차이가 있다고 할 수 있는지 판단하는 것이다.
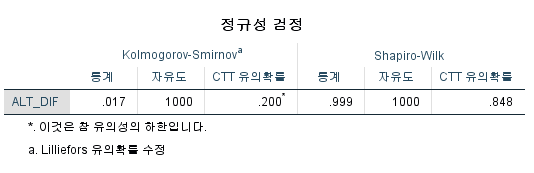
전제: 정규성 검정 (차이)
검정하고자 하는 두 변수의 차이가 정규성을 띤다면 paired t-test를 하면 되므로 정규성 여부를 파악하도록 한다. 따라서 GGT와 간 기능 개선제 복용 후 GGT(GGT_POSTMED)의 차이를 구하고 정규성 검정을 시행한다.
차이를 구하는 방법:
2022.11.30 - [통계 프로그램 사용 방법/SPSS] - [SPSS] 변수 계산 (산술 연산)
정규성 검정을 하는 방법:
2022.08.11 - [기술 통계/SPSS] - [SPSS] 정규성 검정
2022.08.18 - [기술 통계/SPSS] - [SPSS] 고급 Q-Q Plot - Van der Waerden, Rankit, Tukey, Blom
차이 구하기
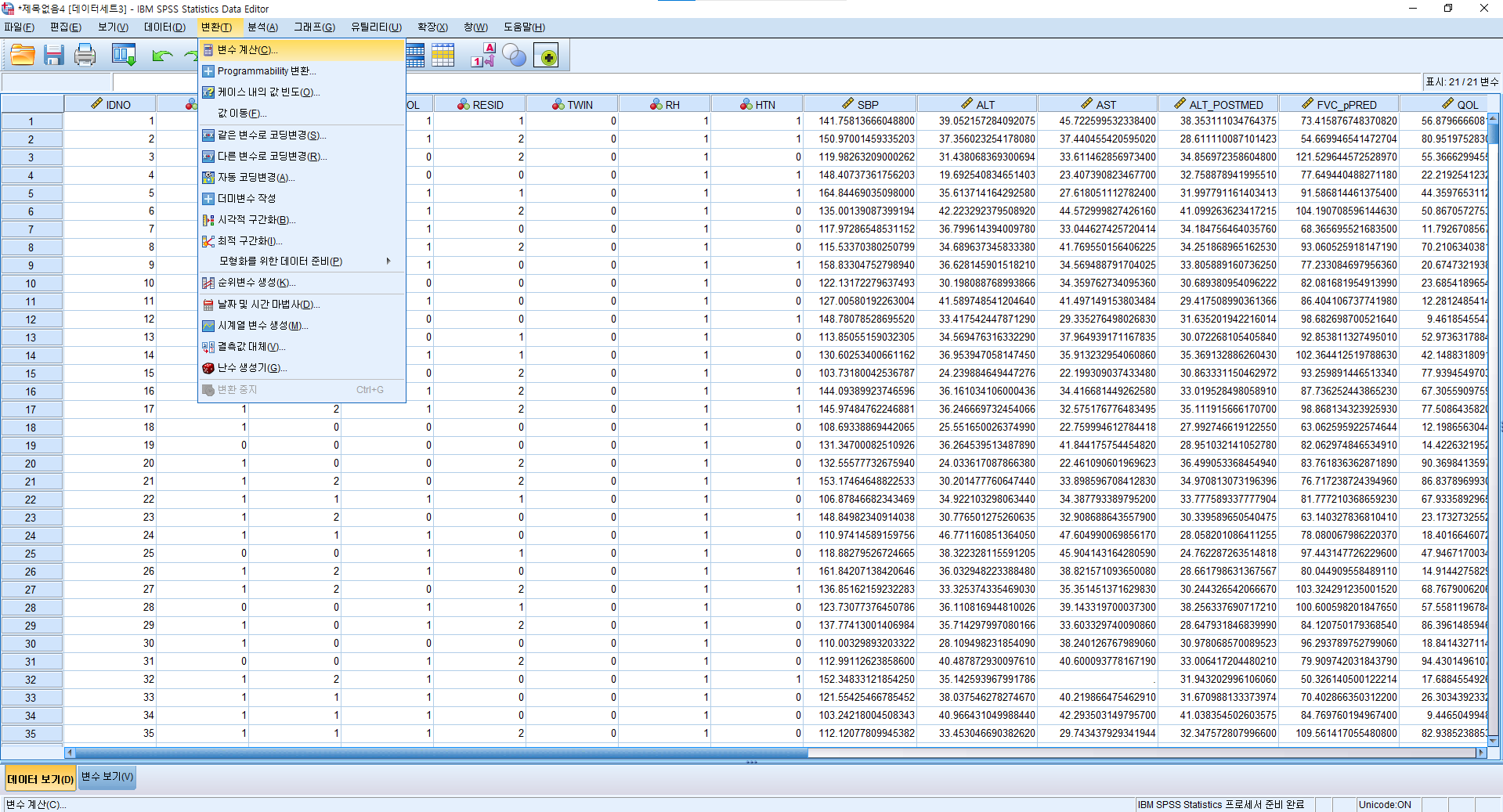
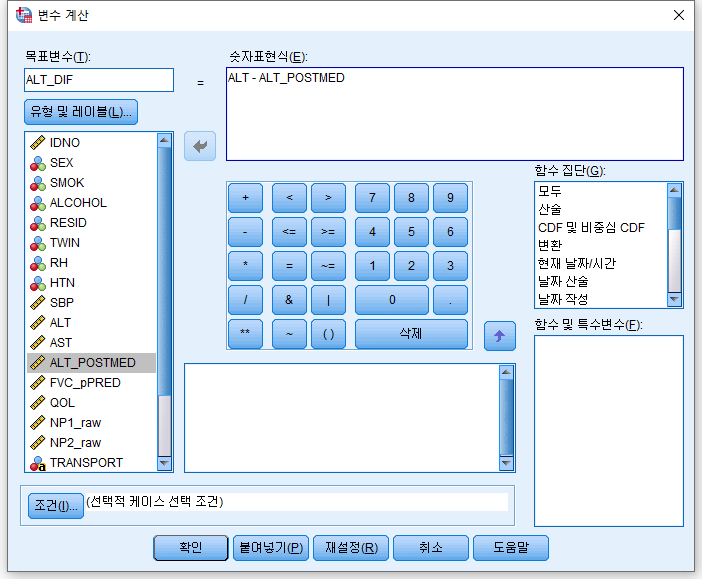
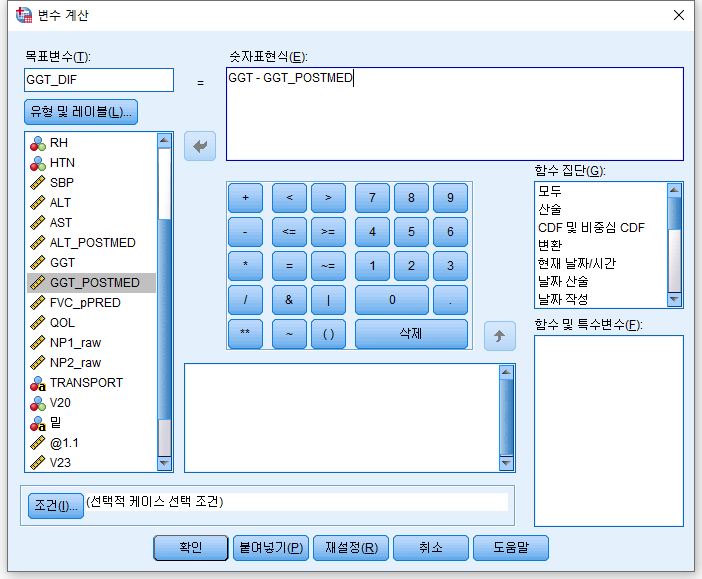
1) 변환(T) > 변수 계산(C)

2) GGT에서 GGT_POSTMED를 빼고 그것을 GGT_DIF에 저장한다.
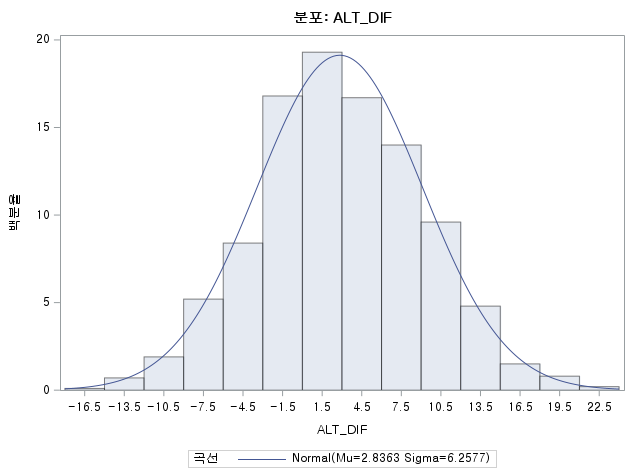
정규성
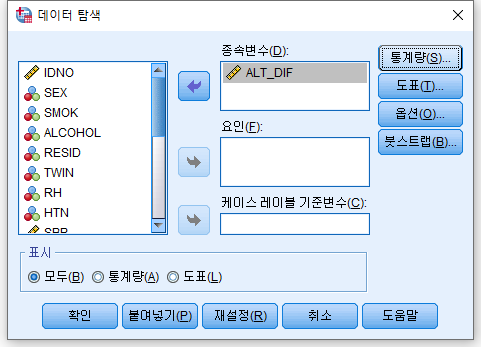
1) 분석(A) > 기술통계량(E) > 데이터 탐색 (E)

2) 분석하고자 하는 변수인 GGT_DIF을 "종속변수"에 넣고, "도표(T)..."를 선택한다.

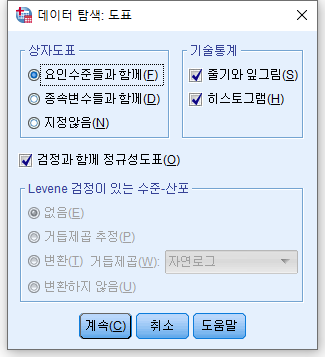
3) "히스토그램(H)", "검정과 함께 정규성 도표(O)" 체크박스를 클릭하고 "계속(C)"를 누르고, 돌아가 "확인"을 누른다.
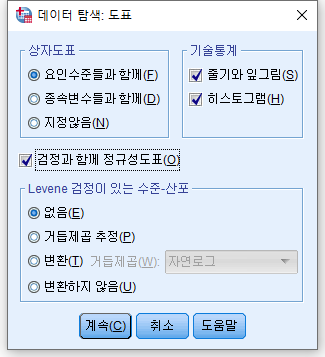
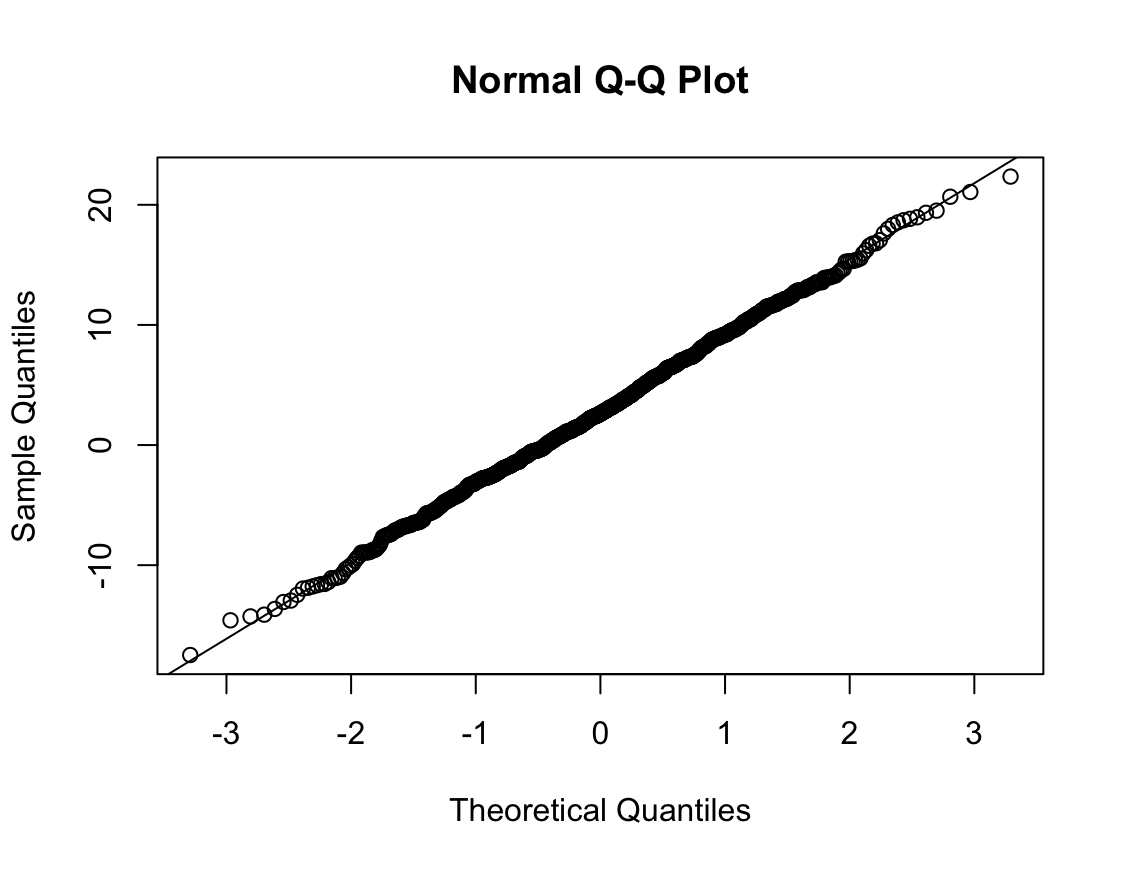
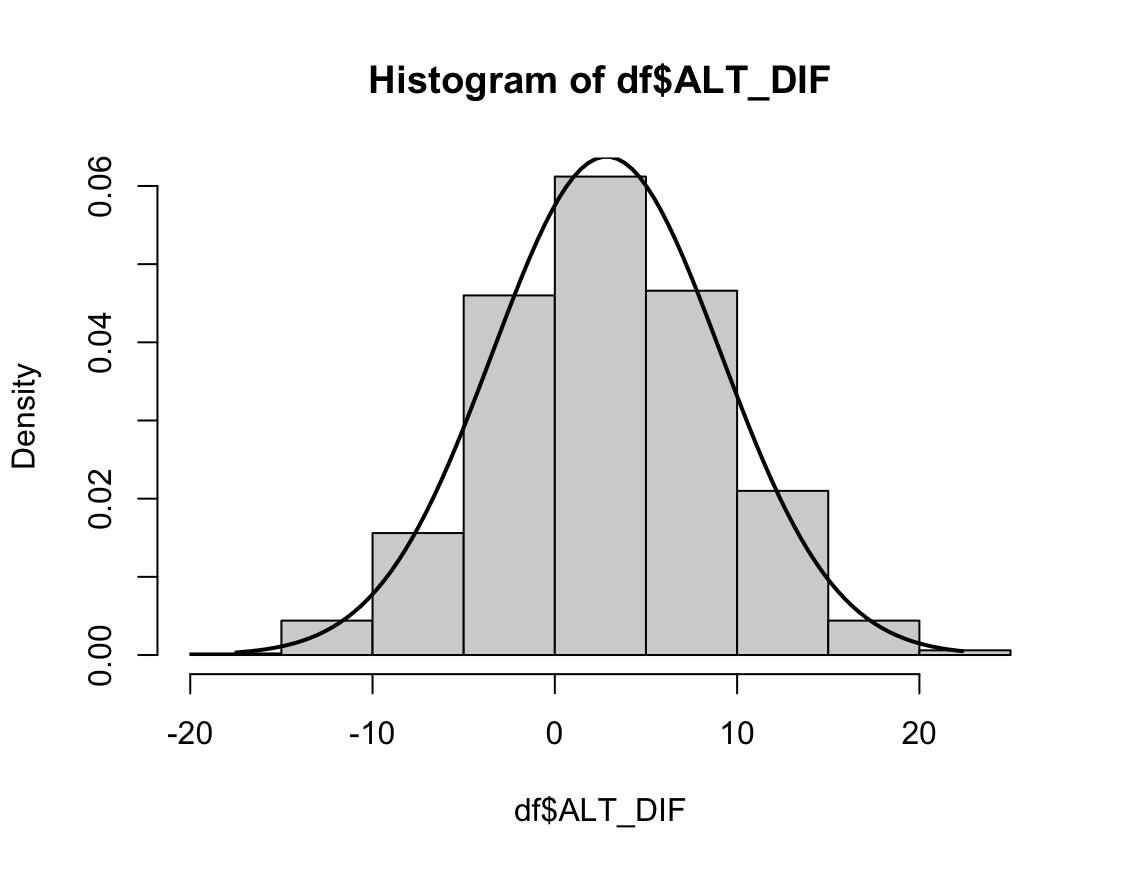
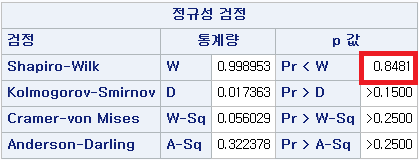
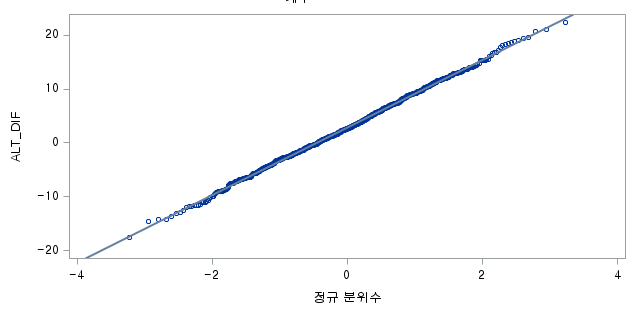
결과

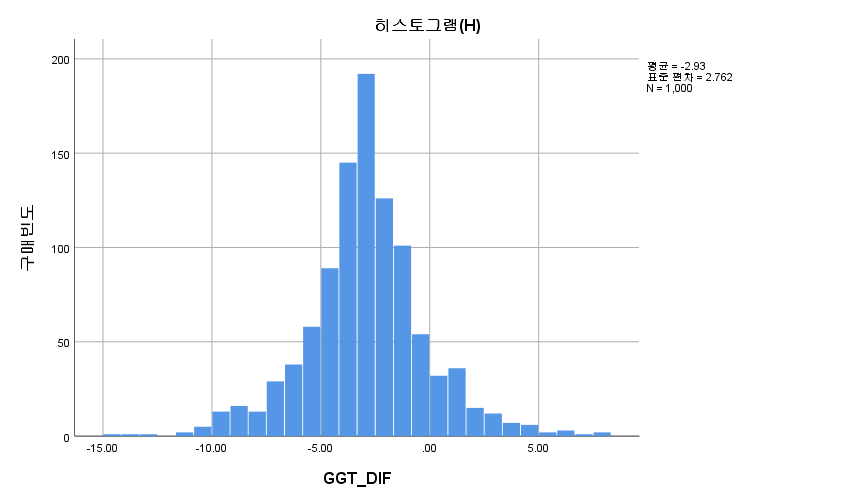

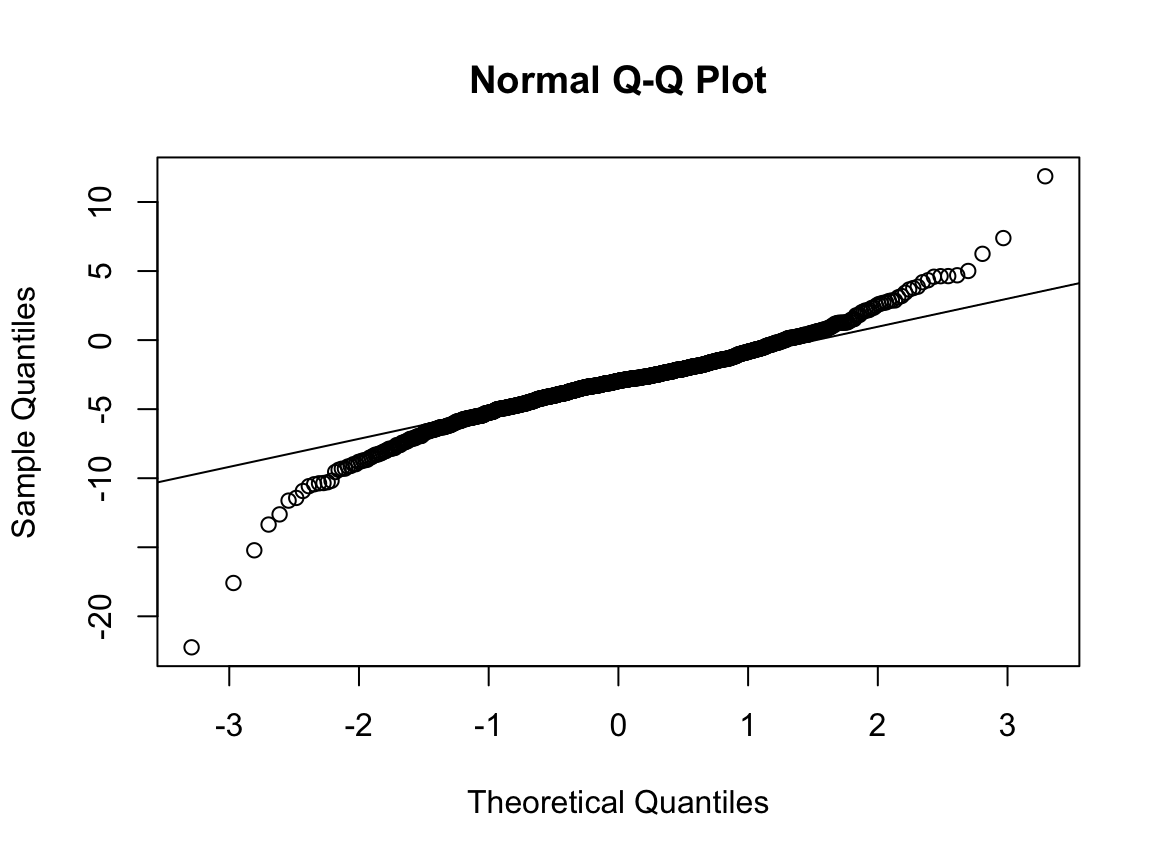
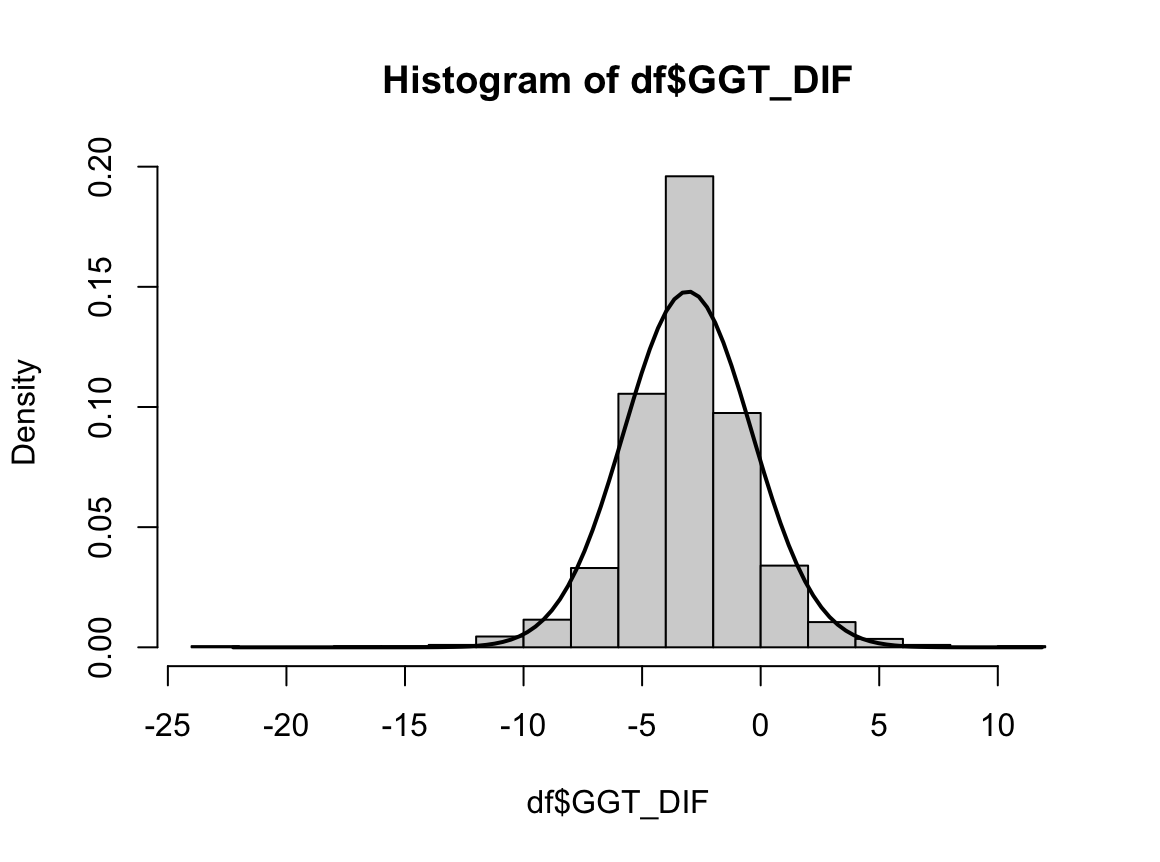
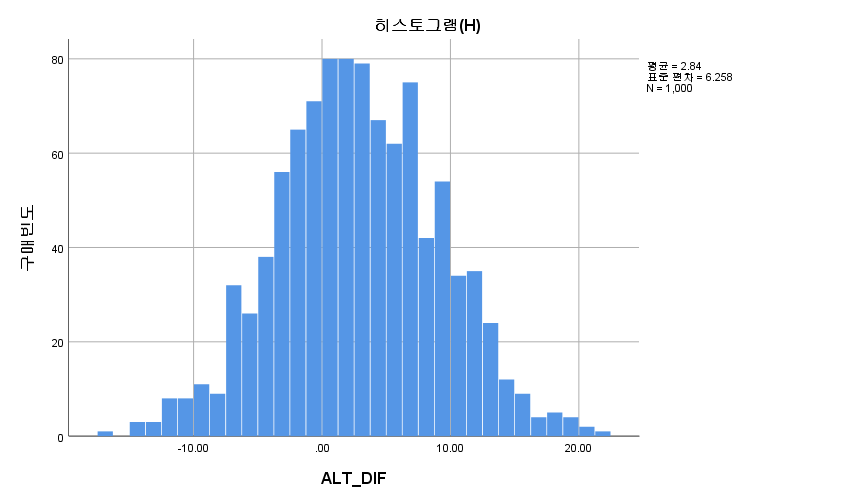
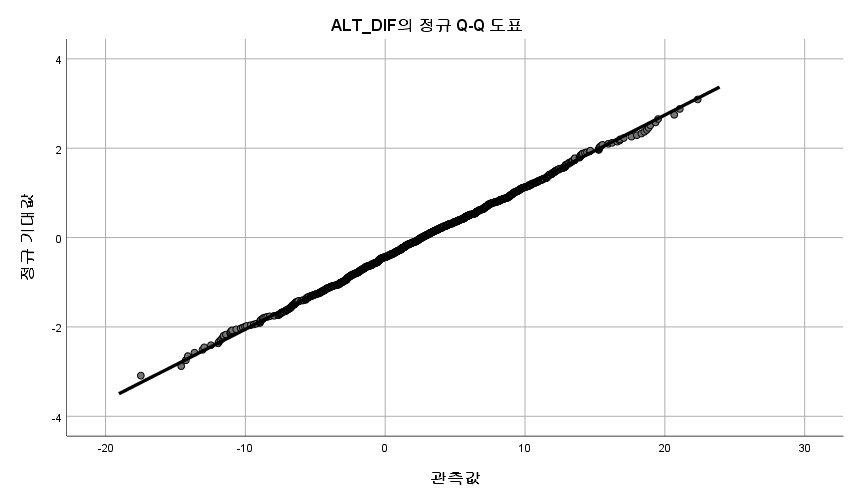
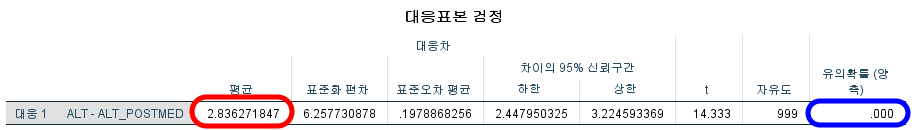
N수가 2,000개 미만이므로 Shapiro-Wilk 통계량의 p-value를 보면 0.05 이하이며, Q-Q Plot은 대부분의 데이터가 선상에 있지 않다. 히스토그램에서는 정규성을 따르는 것처럼 보이지만 이는 개인의 느낌이므로 정확한 것은 아니다. 따라서 대응 표본 T검정 (Paired sample T-test)를 시행할 수 없고, 윌콕슨 부호 순위 검정 (Wilcoxon Signed Rank Test)을 시행해야 한다.
윌콕슨 부호 순위 검정 (Wilcoxon Signed Rank Test)
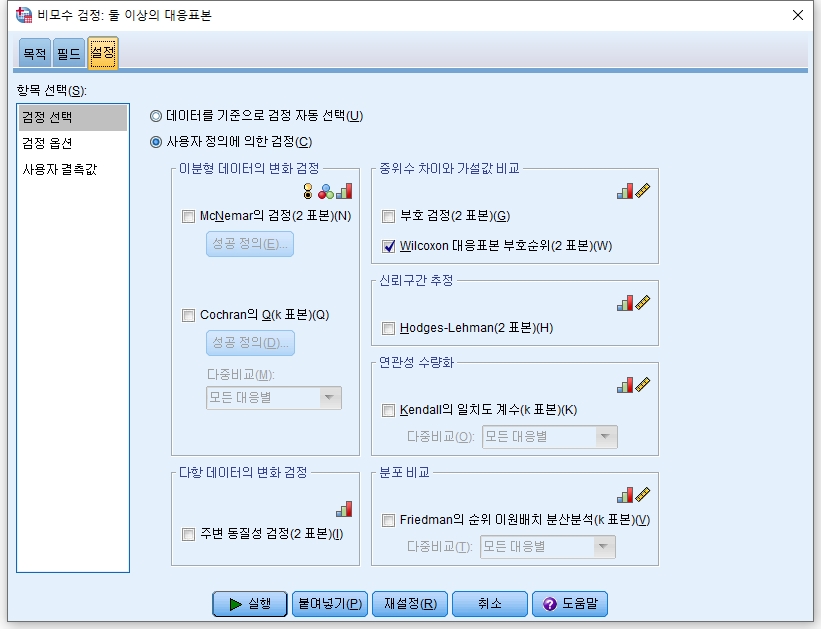
1) 분석(A) > 비모수검정(N) > 대응표본(R)
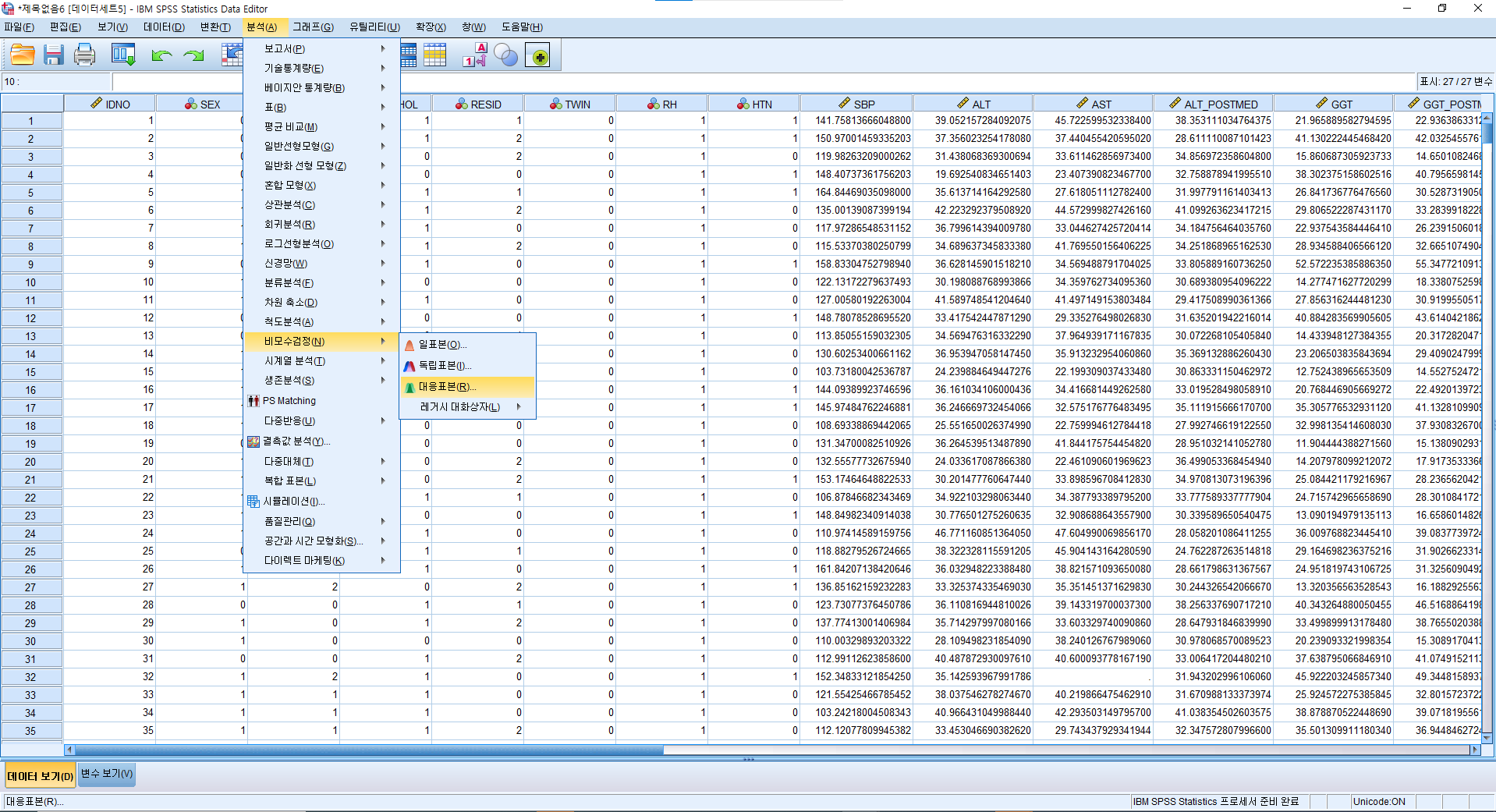
2) 이때 나오는 창의 첫 페이지인 "목적"은 건들지 않는다.

3) "필드"를 누르고 분석하고자 하는 GGT와 GGT_POSTMED를 오른쪽으로 넘긴다.
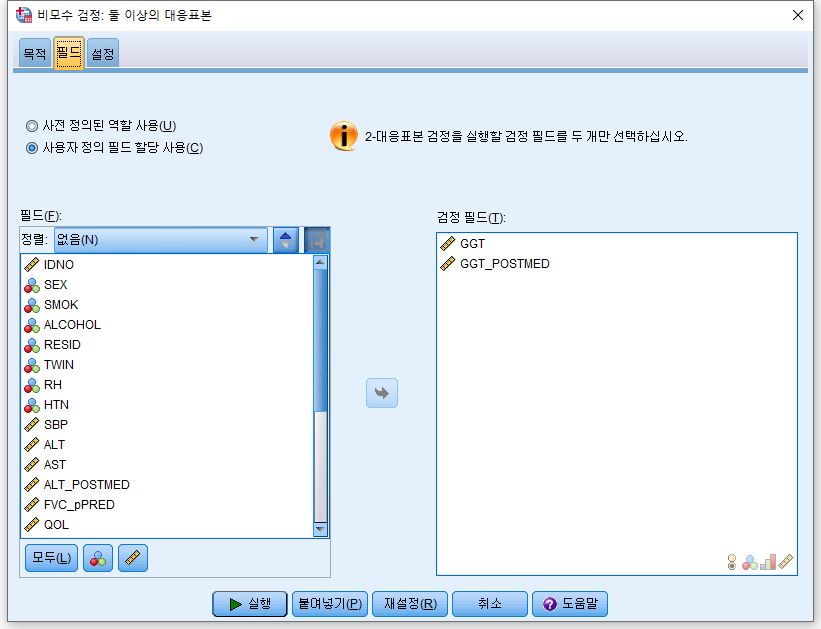
4) "사용자 정의에 의한 검정(C)"를 누르고 "Wilcoxon 대응표본 부호순위(2 표본)(W)"을 체크하고 "실행"을 누른다.
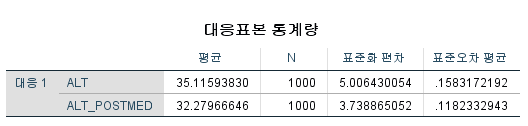
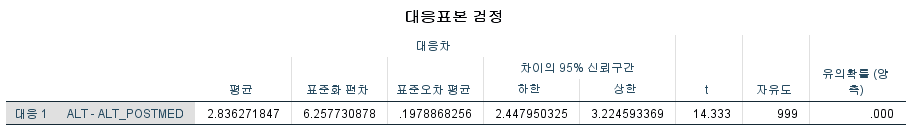
결과

중요한 건 p-value다. 유의확률인 p-value가 0.000(<0.001)로 0.05보다 작으므로 GGT와 GGT_POSTMED는 차이가 있다고 할 수 있다. 지난번 일표본 윌콕슨 부호 순위 검정에서도 밝혔듯이 윌콕슨 부호 순위 검정은 가정이 필요 없는 검정이 아니다. 대칭이라는 가정이 필요하다.(2022.12.02 - [모평균 검정/SPSS] - [SPSS] 일표본 윌콕슨 부호 순위 검정 (비모수 일표본 중앙값 검정: One-Sample Wilcoxon Signed Rank Test)) 따라서 대칭 여부를 확인해야 하는데, 위 GGT_DIF의 히스토그램을 보면 좌우대칭임을 확인할 수 있다. 따라서 원 결론 그대로 차이가 난다고 결론을 내리면 된다.
그런데, 눈치를 챈 독자도 있겠지만, 비모수 일표본 검정도, 대응 표본 검정도 모두 윌콕슨 부호 순위 검정을 실시한다. 이는 모수적인 방법에서 대응 표본 T 검정 (paired T test)가 사실 일표본 T 검정 (One sample T test)와 같다는 것과 일맥상통하는 이야기다. 이 말을 비틀어 생각하면, 일표본 윌콕슨 부호 순위 검정으로 대응표본 윌콕슨 부호 순위 검정을 시행할 수 있다는 말이다. 즉 위에서 만들 DDT_DIF변수로 0에 대해 일표본 윌콕슨 부호 순위 검정을 시행하면 같은 결과를 내는 것을 확인할 수 있다.
1) 분석(A) > 비모수검정(N) > 일표본(O)

2) 이때 나오는 창의 첫 페이지인 "목적"은 건들지 않는다.

3) "필드"를 누르고 분석하고자 하는 GGT_DIF를 오른쪽으로 넘긴다.

4) "사용자 정의에 의한 검정(T)"를 누르고 "평균과 가설값 비교(Wilcoxon 부호 순위 검정)"을 체크하고, 가설 중위수에는 검정하고자 하는 값인 0을 적는다. 그리고 "실행"을 누른다.

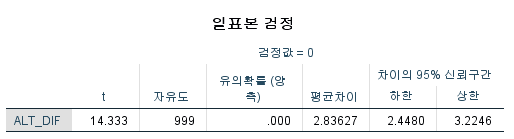
결과

위와 같은 결론을 내리고 있다.
[SPSS] 윌콕슨 부호 순위 검정 (비모수 짝지어진 표본 중앙값 검정: Wilcoxon signed rank test) 정복 완료!
작성일: 2022.12.06.
최종 수정일: 2022.12.06.
이용 프로그램: IBM SPSS v26
운영체제: Windows 10
'반복 측정 자료 분석 > SPSS' 카테고리의 다른 글
| [SPSS] 대응 표본 T검정 (Paired samples T-test) (0) | 2022.11.30 |
|---|